Folding@home : Aider la recherche en mettant à contribution sa puissance de calcul

Le 06/04/2020

4 minutes de lecture
Folding@home : Aider la recherche en mettant à contribution sa puissance de calcul
En cette période de pandémie de COVID-19, beaucoup d’espoir se porte sur la recherche, notamment pour le développement d'un vaccin. L'étude du virus passe entre autres par la simulation de différents modèles de
repliement des protéines.
Traditionnellement, ces simulations très coûteuses en puissance de calcul se font par des superordinateurs, tel que IBM Blue Gene :

Une autre méthode, mise au point par l'université de Standford en 2000, consiste à agréger la puissance de calcul des ordinateurs des particuliers à travers le monde en un immense cluster de calcul distribué. Il s'agit du projet
Folding@home (FAH).
Chacun peut y participer pour faire avancer la recherche sur le cancer, les maladie d'Alzheimer, Parkinson et Huntington, et plus récemment pour le nouveau coronavirus 2019-nCoV.
Nous allons nous intéresser ici à l'installation du client FAH pour participer à cet effort commun.
1. Installation
Commencer par télécharger le programme d'installation du client depuis
la page du projet.
Le logiciel est disponible sur Windows, Mac, Linux et Android.
Procéder à l'installation en mode "express".
2. Utilisation
Suite à l'installation, le client se lance en arrière-plan et une page Web d'administration s'affiche.
Depuis cette page, il est possible de configurer quelques paramètres :
- Power : La proportion de ressources CPU et/ou GPU qui sera utilisée, configurable à Light, Medium ou Full.
- Le type de recherche à supporter. Pour la recherche sur le COVID-19, laisser l'option par défaut "Any disease". Il est aussi possible de rejoindre d'autres recherches spécifiques.
- La modalité de lancement des traitements, soit tout le temps "While I'm working" ou bien seulement pendant les périodes d'inactivité de l'ordinateur "Only when I'm idle".
- L'identité : Par défaut l'ordinateur va travailler de manière anonyme et autonome, mais il est possible de saisir un pseudonyme et de rejoindre une équipe. L'équipe gagne alors des points
virtuels attribués pour l'accomplissement de tâches (Work Units) et participe à une forme symbolique de compétition. Le classement des équipes est disponible sur la page du projet.
On peut voir ici que le CPU de la machine travaille sur un des
projets lié au coronavirus :
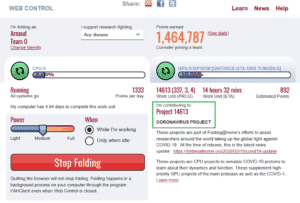
Après avoir configuré le client, les premières Work Units vont être traitées en arrière-plan. Il est alors possible de fermer la console web, le client continuera de fonctionner en arrière-plan.
Attention, si l'on configure le client sur l'option Power à "Full", le CPU et/ou le GPU de la machine vont être beaucoup sollicités, bien que le logiciel évite l'utilisation de 100% des ressources. Ce mode est à réserver sur des PC fixes munis d'un système de refroidissement un minimum performant, et à exclure sur les laptops qui risquent la surchauffe.
Enfin, il faut bien avoir à l'esprit que laisser fonctionner les simulations sur de longues périodes a un coût sur la facture d’électricité. Il faut donc voir ce projet comme une manière alternative et originale de faire un don pour la recherche.
3. Portée du projet
Les CPU et GPU de nos ordinateurs sont souvent peu performants et sans comparaison avec un superordinateur, cependant l'union fait la force, car depuis l'annonce de projets pour la recherche COVID-19, la capacité cumulée de calcul du réseau est passée à
1,5 ExaFLOPS comme on peut le voir sur la
page de statistiques du projet.
A titre de comparaison, une initiative d'IBM visant à faire travailler 16 superordinateurs, entre autres opérés par le MIT et la NASA, atteint une capacité cumulée de "seulement"
0,33 ExaFLOPs.
Folding@home est donc actuellement très important pour la recherche.
 Une autre méthode, mise au point par l'université de Standford en 2000, consiste à agréger la puissance de calcul des ordinateurs des particuliers à travers le monde en un immense cluster de calcul distribué. Il s'agit du projet Folding@home (FAH).
Chacun peut y participer pour faire avancer la recherche sur le cancer, les maladie d'Alzheimer, Parkinson et Huntington, et plus récemment pour le nouveau coronavirus 2019-nCoV.
Nous allons nous intéresser ici à l'installation du client FAH pour participer à cet effort commun.
Une autre méthode, mise au point par l'université de Standford en 2000, consiste à agréger la puissance de calcul des ordinateurs des particuliers à travers le monde en un immense cluster de calcul distribué. Il s'agit du projet Folding@home (FAH).
Chacun peut y participer pour faire avancer la recherche sur le cancer, les maladie d'Alzheimer, Parkinson et Huntington, et plus récemment pour le nouveau coronavirus 2019-nCoV.
Nous allons nous intéresser ici à l'installation du client FAH pour participer à cet effort commun.
 Après avoir configuré le client, les premières Work Units vont être traitées en arrière-plan. Il est alors possible de fermer la console web, le client continuera de fonctionner en arrière-plan.
Attention, si l'on configure le client sur l'option Power à "Full", le CPU et/ou le GPU de la machine vont être beaucoup sollicités, bien que le logiciel évite l'utilisation de 100% des ressources. Ce mode est à réserver sur des PC fixes munis d'un système de refroidissement un minimum performant, et à exclure sur les laptops qui risquent la surchauffe.
Enfin, il faut bien avoir à l'esprit que laisser fonctionner les simulations sur de longues périodes a un coût sur la facture d’électricité. Il faut donc voir ce projet comme une manière alternative et originale de faire un don pour la recherche.
Après avoir configuré le client, les premières Work Units vont être traitées en arrière-plan. Il est alors possible de fermer la console web, le client continuera de fonctionner en arrière-plan.
Attention, si l'on configure le client sur l'option Power à "Full", le CPU et/ou le GPU de la machine vont être beaucoup sollicités, bien que le logiciel évite l'utilisation de 100% des ressources. Ce mode est à réserver sur des PC fixes munis d'un système de refroidissement un minimum performant, et à exclure sur les laptops qui risquent la surchauffe.
Enfin, il faut bien avoir à l'esprit que laisser fonctionner les simulations sur de longues périodes a un coût sur la facture d’électricité. Il faut donc voir ce projet comme une manière alternative et originale de faire un don pour la recherche.



 Le 06/04/2020
Le 06/04/2020 4 minutes de lecture
4 minutes de lecture




